こんにちは~。ガイです。前回の続きを書いていこうと思います。
前回までで、Qiime2 conceptual-overview でいうところのFeature Table とRepresentative Sequences が手に入りました。ここからようやく解析らしい解析に入っていけます。本当はconceptual-overview 貼りたかったんですが、画像引用OK かよくわからなかったのでよかったら以下のページを見てみてください。
- METADATA.tsv の作成
- Taxonomy analysis
METADATA.tsv の作成
今後、様々な解析に使うメタデータを作成します。とっても大事です。
sample-id を一番左に置き、ほかの列に採集部位や群名など、今後の解析におけるグループ分けに必要な情報などを入れていきます。自分のデータに合わせてExcel などで作ってください。
Excel ファイルをtsv 形式で保存するには、テキスト(タブ形式)で保存し、拡張子を.tsv に直します。
ちなみに、公式が公開しているメタデータは以下のような内容です。
参考:https://data.qiime2.org/2023.2/tutorials/moving-pictures/
Taxonomy analysis
RESCRIPt を使ってNaive Bayes 分類器を作成
Qiime2 による分類には、まずNaive Bayes 分類器を作る必要があります。
一応、すでにトレーニングされたデータベースも公開されているんですが、公式が以下のように言っておられます。
Taxonomic classifiers perform best when they are trained based on your specific sample preparation and sequencing parameters, including the primers that were used for amplification and the length of your sequence reads. Therefore in general you should follow the instructions in Training feature classifiers with q2-feature-classifier to train your own taxonomic classifiers (for example, from the marker gene reference databases below). Data resources — QIIME 2 2023.2.0 documentation
よって、以下のチュートリアルを参考に進めました。
ちなみに、V3-V4 領域だけをトレーニングすることもできるようなのですが、試していません。今回、特殊なプライマーを使用したこともあり、フルレングスで進めています。
RESCRIPt および周辺ソフトのインストール
初めに、公式の指示にしたがってインストールを試みました。
公式:GitHub - bokulich-lab/RESCRIPt: REference Sequence annotation and CuRatIon Pipeline
conda install \ -c conda-forge -c bioconda -c qiime2 -c https://packages.qiime2.org/qiime2/2023.5/tested/ -c defaults \ qiime2 q2cli q2templates q2-types q2-longitudinal q2-feature-classifier 'q2-types-genomics>2023.2' \ "pandas>=0.25.3" xmltodict ncbi-datasets-pylib
これでインストールできた人は以下は読み飛ばしてかまいません。わたしは上手くいかなかったため、公式に書かれていたソフトウェアをひとつひとつインストールしていくことにしました。
conda install -c conda-forge xmltodict
conda install -c bioconda ncbi-datasets-pylib
conda install -c https://packages.qiime2.org/qiime2/2023.5/tested/ 'q2-types-genomics>2023.5' #これが全然進まない。
ModuleNotFoundError: No module named 'q2_types_genomics' のトラブルシューティング
conda install -c https://packages.qiime2.org/qiime2/2023.5/tested/ 'q2-types-genomics>2023.5' が一生終わらないので業を煮やしてRESCRIPtのインストールをやってみました。すると
pip install git+https://github.com/bokulich-lab/RESCRIPt.git
#To view a help menu for using rescript via the QIIME 2 CLI: qiime dev refresh-cache ModuleNotFoundError: No module named 'q2_types_genomics'
やはりq2_types_genomics がないと先に進めないようです。しかも、さらに悪いことに、無理やり入れようとしたせいなのかQiime2 そのものが動かなくなる悪夢に見舞われました。こうなったらどうにかしてq2_types_genomics を入れるしかない。
公式スクリプトを見るに、https://packages.qiime2.org/qiime2/2023.5/tested/ に何かあるだろうと思ったので、とんでみるとlinux-64 というページがありました。

そのページになんとなくよさげな以下のファイル(https://packages.qiime2.org/qiime2/2023.5/tested/linux-64/q2-types-genomics-2023.5.0.dev0+4.gc4f304a-py38_0.tar.bz2)を発見したので、最新作をwget でget。
wget https://packages.qiime2.org/qiime2/2023.5/tested/linux-64/q2-types-genomics-2023.5.0.dev0+4.gc4f304a-py38_0.tar.bz2
解凍してみます。
tar -jxvf q2-types-genomics-2023.5.0.dev0+4.gc4f304a-py38_0.tar.bz2
「lib」と「info」というフォルダが出てきました。
lib の中を見ると「lib/python3.8/site-packages/lib/python3.8/site-packages/q2_types_genomics」と「q2_types_genomics-2023.5.0.dev0+4.gc4f304a-py3.8.egg-info」というフォルダがありました。
さらに、まったく同じ名前のフォルダをminiconda フォルダの中に発見しました。
/miniconda3/envs/qiime2-2023.2/lib/python3.8/site-packages
ここに先ほどのq2_types_genomicsとq2_types_genomics-2023.5.0.dev0+4.gc4f304a-py3.8.egg-infoを移してみました。
そして、qiime --help を実行するとqiime2 が動くようになっていました。
よかった~。
さらに、 qiime rescript --help
Usage: qiime rescript [OPTIONS] COMMAND [ARGS]...
rescript も動き出しました!
が、危ないかもしれないのでこの方法使う際は自己責任でお願いします!
SILVAデータを取得する
公式チュートリアル:Getting SILVA data the easy way https://forum.qiime2.org/t/processing-filtering-and-evaluating-the-silva-database-and-other-reference-sequence-data-with-rescript/15494
qiime rescript get-silva-data \
--p-version '138.1' \
--p-target 'SSURef_NR99' \
--o-silva-sequences silva-138.1-ssu-nr99-rna-seqs.qza \
--o-silva-taxonomy silva-138.1-ssu-nr99-tax.qza
オプションの説明
##select specific ranks for your SILVA taxonomy
--p-ranks flag to the either the parse-silva-taxonomy or get-silva-data commands.
###For example:
--p-ranks domain kingdom phylum class order suborder family subfamily genus
上手くいくと以下の文章が表示されます。
Saved FeatureData[RNASequence] to: silva-138.1-ssu-nr99-rna-seqs.qza Saved FeatureData[Taxonomy] to: silva-138.1-ssu-nr99-tax.qza
ダウンロードしたRNA 配列をDNA 配列にリバース
この公式チュートリアルでダウンロードした配列はRNA 配列なので、DNA 配列にリバースする必要があります。
公式チュートリアル:https://forum.qiime2.org/t/processing-filtering-and-evaluating-the-silva-database-and-other-reference-sequence-data-with-rescript/15494
qiime rescript reverse-transcribe \
--i-rna-sequences silva-138.1-ssu-nr99-rna-seqs.qza \
--o-dna-sequences silva-138.1-ssu-nr99-seqs.qza
DNA 配列から低品質配列を除去
公式チュートリアル:“Culling” low-quality sequences with cull-seqs Processing, filtering, and evaluating the SILVA database (and other reference sequence data) with RESCRIPt - Tutorials - QIIME 2 Forum
qiime rescript cull-seqs \
--i-sequences silva-138.1-ssu-nr99-seqs.qza \
--o-clean-sequences silva-138.1-ssu-nr99-seqs-cleaned.qza
配列を長さと分類群でフィルタリングする
公式チュートリアル:Filtering sequences by length and taxonomy
qiime rescript filter-seqs-length-by-taxon \
--i-sequences silva-138.1-ssu-nr99-seqs-cleaned.qza \
--i-taxonomy silva-138.1-ssu-nr99-tax.qza \
--p-labels Archaea Bacteria Eukaryota \
--p-min-lens 900 1200 1400 \
--o-filtered-seqs silva-138.1-ssu-nr99-seqs-filt.qza \
--o-discarded-seqs silva-138.1-ssu-nr99-seqs-discard.qza
重複配列の除去
公式チュートリアル:Dereplication of sequences and taxonomy
メモリ128GB で一時間かかったというメモが残されており、かなり重い作業と思われますのでご注意。わたしはスパコンを使いました。
qiime rescript dereplicate \
--i-sequences silva-138.1-ssu-nr99-seqs-filt.qza \
--i-taxa silva-138.1-ssu-nr99-tax.qza \
--p-perc-identity 0.97 \
--p-mode 'lca' \
--o-dereplicated-sequences silva-138.1-ssu-c97-seqs-derep-lca.qza \
--o-dereplicated-taxa silva-138.1-ssu-c97-tax-derep-lca.qza
Naive Bayes 分類器(classifier-naive-bayes)の作成
公式チュートリアル:![]() Wonderful!
Wonderful! ![]()
Now we are ready to make our classifier for use on full-length SSU sequences. ![]()
これメモリ64GB で四時間かかったというメモが残されていたのでご注意ください。
qiime feature-classifier fit-classifier-naive-bayes \
--i-reference-reads silva-138.1-ssu-c97-seqs-derep-lca.qza \
--i-reference-taxonomy silva-138.1-ssu-c97-tax-derep-lca.qza \
--o-classifier silva-138.1-ssu-nr99-classifier.qza
これにて分類器のできあがり。
Naive Bayes 分類器を使ってサンプルの分類学的構成を調べる
公式チュートリアル:“Moving Pictures” tutorial — QIIME 2 2020.8.0 documentation
qiime feature-classifier classify-sklearn \
--i-classifier ~/path/to/silva-138.1-ssu-nr99-classifier.qza \
--i-reads qza/rep-seqs.qza \
--o-classification qza/taxonomy.qza
feature-classifier classify-sklearn の結果の可視化
qiime metadata tabulate \
--m-input-file qza/taxonomy.qza \
--o-visualization qzv/taxonomy.qzv
qzv ファイルをQiime2 View https://view.qiime2.org/に投げ込みます。

ここまでのアウトプットの整理
table.qza # FeatureTable[Frequency]: 配列ID(ASV)ごとに各サンプルにおける頻度が格納されている
stats.qza # SampleData[DADA2Stats]: DADA2 後に残ったリードの頻度情報
rep-seqs.qza # FeatureData[Sequence]: 配列ID と配列情報
例 >4b5eeb300368260019c1fbc7a3c718fc TACGGAGGATCCGAGCGTTATCCGGATTTATTGGGTTTAAAGGGAGCGTAGATGGATGTTTAAGTCAGTTGTGAAAGTTTGCGGCTCAACCGTAAAATTGCAGTTGATACTGGATATCTT
taxonomy.qza # FeatureData[Taxonomy]: 配列ID と分類群情報とその頻度
Feature ID #q2:types Taxon categorical Confidence categorical ff8c7f94f941a0c647120d4e142db316 k__Bacteria; p__Firmicutes; c__Clostridia; o__Clostridiales; f__Veillonellaceae; g__Dialister; s__ 0.9997197443199501
ff3df50eea7490dd794148649c480fd3 k__Bacteria; p__Firmicutes; c__Clostridia; o__Clostridiales; f__Lachnospiraceae 0.9920784845661842
菌叢の可視化(棒グラフの作成)
qiime taxa barplot \
--i-table qza/selected_table.dada.qza \
--i-taxonomy qza/taxonomy.qza \
--m-metadata-file METADATA.tsv \
--o-visualization qzv/taxa-bar-plots.qzv
qzv ファイルをQiime2 View https://view.qiime2.org/に投げ込むと以下のようなグラフが出てきます。
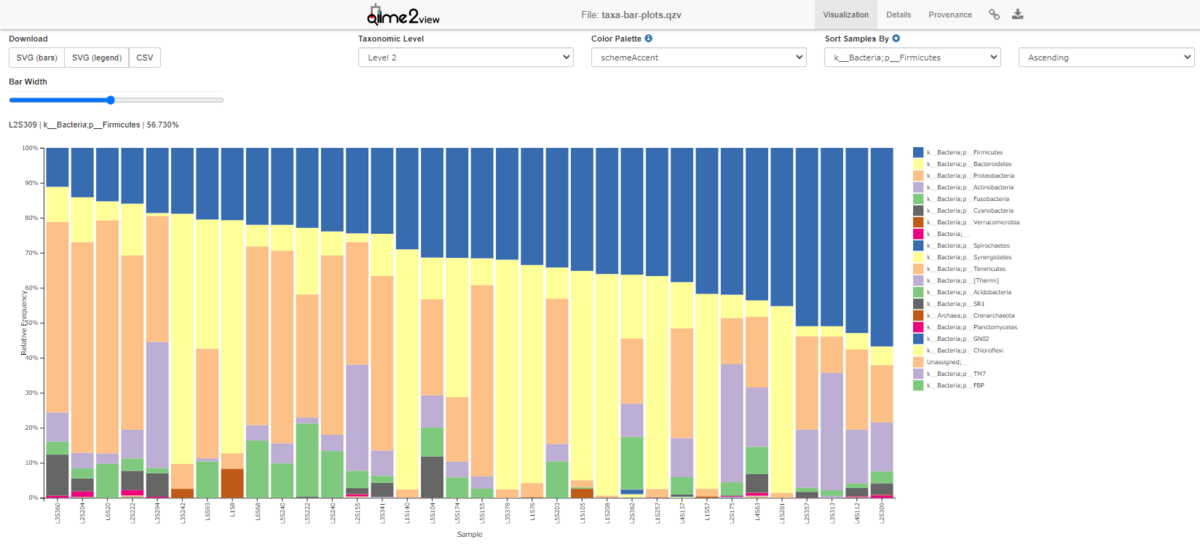
Taxonomic Level を変更することで、Level1のKingdom:界からLevel7の種まで分類レベルを変更できます。上の図ではLevelは2になっています。
Sort Samples By を変更することで棒グラフ整列時の優先順を決めることができます。 以下の写真では分類群を種(Level 7)まで分類し、優先順位をbody-site で決めています。

今日のところはここまでにしておきます!お疲れ様でした。
